R Markdown
Este es un documento en formato R Markdown. Markdown es un sintaxis de formato simple para crear documentos HTML, PDF, y MS Word. Para más detalles de como utilizar R Markdown visita http://rmarkdown.rstudio.com.
Cuando presoinas el botón Knit, se genera un documento que incluye tanto el contenido como el como el resultado de cualquier trozo de codigo R incrustado dentro del documento. Se puede incrustar un trozo de codigo R así:
Carga de paquetes necesarios
El siguiente codigo carga los paquetes:
- readr:
- mosaic:
- FSA:
- ggplot2:
- readxl:
- knitr:
- doBy:
- agricolae:
library(readr)
library(mosaic)
library(FSA)
library("ggplot2")
library(readxl)
library(knitr)
library(doBy)
library(agricolae)
options(scipen=999, digits = 0)
## Load all the libraries needed and the dataCarga de archivos necesarios
El siguiente codigo carga los archivos:
- read_excel:
mydata <- read_excel("./data/2016-Solanum-01-11_Isolates(Germplasms).xlsx")Manejo de datos
Para manipular de manera eficiente los datos de nuestro archivo, es conveniente realizar subconjuntos de datos con el comando Subset. Con el siguiente codigo seleccionamos las variables independientes y de respuesta que analizaremos posteriormente.
- Subset:
####Eggs PER PLANT ####
Sub.Eggs <- subset(mydata,
select=c(Nger,
Isolate,
EggsP) )
##Create a subset of the desire variableEstadística descriptiva
Para este ejemplo nuestro objetivo es saber si las variables germoplasma (Nger) y aislado (Isolate) influyen significativamente sobre la variable numero de huevos por planta (EggsP). El siguiente método ilustra de manera sencilla como calcular y almacenar las respuestas necesarias para realizar un reporte de resultados.
En principio, nuestro conjunto de datos se compone de 5 tratamientos en germoplasma y 11 en aislados. Esto nos permite realizar nuestro análisis desde dos enfoques: intentar conocer si existe diferencia significativa entre el numero de huevos de cada aislado que se reprodujo en cada germoplasma, o si un mismo aislado se reproduce de manera diferente en cada uno de los germoplasmas. En este post nos enfocaremos en el primero, el analisis de cuatro tratamientos, germoplasmas, para 11 aislados diferentes.
Dado que nuestro conjunto de datos contiene una etiqueta especifica para cada tratamiento, podemos hacer buen uso de Copiar y Pegar, y Buscar y Reemplazar, para ahorrarnos un poco de trabajo. En este caso, solo necesitamos realizar el analisis para un aislado, y para el resto de ellos solo reemplazar su etiqueta por la del siguiente aislado. En futuras entradas se describirá como automatizar este proceso.
Manejo de datos
Para obtener los datos de nuestro primer aislado utilizamos nuevamente el comando subset, al cual le añadimos el parametro Isolate == “MIAd” para seleccionar solo los datos asociados a la etiqueta MIAd en la columna Isolate, y seleccionamos los datos de la columna Nger y EggsP. La siguiente linea indica que los valores de la columna Nger sean tratados como factores.
####Isolate: MIAd####
Sub.Eggs.MIAd <- subset(Sub.Eggs, Isolate == "MIAd", select=c(Nger, EggsP))
Sub.Eggs.MIAd$Nger = factor (Sub.Eggs.MIAd$Nger, labels=c("EC", "TB", "TE", "TS", "TT")) Promedios y error estándar
Para reportar resultados es necesario conocer el promedio y el error estandar de cada uno de los germoplasmas. Para ello, las primeras 5 lineas de codigo se repiten para cada tratamiento. Primero se obtiene el subconjunto del tratamiento y se almacena en la variable. A partir de esta variable se calculan el promedio y el error estandar por medio de los comandos mean y se, del paquete ___, se almacenan en su propia variable, y se redondea el valor a dos digitos (round).
###Descriptive statistics calculation###
Sub.Eggs.MIAd.EC <- subset(Sub.Eggs.MIAd, Nger == "EC", select=c(Nger,EggsP))
Me.EP.MIAd.EC <- mean(Sub.Eggs.MIAd.EC$EggsP, na.mr=FALSE)
Me.EP.MIAd.EC <- round(Me.EP.MIAd.EC, digits=2)
Se.EP.MIAd.EC <- se(Sub.Eggs.MIAd.EC$EggsP)
Se.EP.MIAd.EC <- round(Se.EP.MIAd.EC, digits=2)
Sub.Eggs.MIAd.SB <- subset(Sub.Eggs.MIAd, Nger == "TB", select=c(Nger,EggsP))
Me.EP.MIAd.TB <- mean(Sub.Eggs.MIAd.SB$EggsP, na.mr=FALSE)
Me.EP.MIAd.TB <- round(Me.EP.MIAd.TB, digits=2)
Se.EP.MIAd.TB <- se(Sub.Eggs.MIAd.SB$EggsP)
Se.EP.MIAd.TB <- round(Se.EP.MIAd.TB, digits=2)
Sub.Eggs.MIAd.SB <- subset(Sub.Eggs.MIAd, Nger == "TE", select=c(Nger,EggsP))
Me.EP.MIAd.TE <- mean(Sub.Eggs.MIAd.SB$EggsP, na.mr=FALSE)
Me.EP.MIAd.TE <- round(Me.EP.MIAd.TE, digits=2)
Se.EP.MIAd.TE <- se(Sub.Eggs.MIAd.SB$EggsP)
Se.EP.MIAd.TE <- round(Se.EP.MIAd.TE, digits=2)
Sub.Eggs.MIAd.SB <- subset(Sub.Eggs.MIAd, Nger == "TS", select=c(Nger,EggsP))
Me.EP.MIAd.TS <- mean(Sub.Eggs.MIAd.SB$EggsP, na.mr=FALSE)
Me.EP.MIAd.TS <- round(Me.EP.MIAd.TS, digits=2)
Se.EP.MIAd.TS <- se(Sub.Eggs.MIAd.SB$EggsP)
Se.EP.MIAd.TS <- round(Se.EP.MIAd.TS, digits=2)
Sub.Eggs.MIAd.SB <- subset(Sub.Eggs.MIAd, Nger == "TT", select=c(Nger,EggsP))
Me.EP.MIAd.TT <- mean(Sub.Eggs.MIAd.SB$EggsP, na.mr=FALSE)
Me.EP.MIAd.TT <- round(Me.EP.MIAd.TT, digits=2)
Se.EP.MIAd.TT <- se(Sub.Eggs.MIAd.SB$EggsP)
Se.EP.MIAd.TT <- round(Se.EP.MIAd.TT, digits=2)Kruskal–Wallis test
Una vez obtenidos los valores de promedios y error estandar, podemos dejarlos de lado y proceder a las pruebas estadisticas. Dado que se sabe que los datos no pertenecen a una distribucion normal, el analisis no-paramétrico Kruskall-Wallis se calcula utilizando la función kruskal.test del paquete ____ (las pruebas de normalidad las pondré en otra entrada). En este caso, el resultado se guarda en la variable KTMIAd, que será útil más adelante. Para comprender mejor porqué, se llama a la variable para ver que resultado arroja. Se observa que tenemos un valor de P igual a 0.00007, existe diferencia significativa entre los tratamientos. La funcion ls “devuelve un vector de cadenas de caracteres que dan los nombres de los objetos en el entorno especificado”, y con ella podemos ver que el valor de P se encuentra almacenado bajo el nombre de p.value y podemos llamarlo mediante variable$nombre.
####Kruskal-Wallis test calculation####
KTMIAd = kruskal.test(Sub.Eggs.MIAd$EggsP ~ Sub.Eggs.MIAd$Nger)
KTMIAd##
## Kruskal-Wallis rank sum test
##
## data: Sub.Eggs.MIAd$EggsP by Sub.Eggs.MIAd$Nger
## Kruskal-Wallis chi-squared = 30, df = 4, p-value = 0.000007ls(KTMIAd)## [1] "data.name" "method" "p.value" "parameter" "statistic"KTMIAd$p.value## [1] 0Dunn’s test
La prueba anterior indica si existe, o no, diferencia entre los tratamientos. Para hacer una comparación entre los tratamientos se utiliza la función dunnTest.
####Dunn test calculation####
DTMIAd = dunnTest(Sub.Eggs.MIAd$EggsP ~ Sub.Eggs.MIAd$Nger, method="bonferroni")
ls(DTMIAd)## [1] "dtres" "method" "res"DTMIAd$res## Comparison Z P.unadj P.adj
## 1 EC - TB 5 0 0
## 2 EC - TE 3 0 0
## 3 TB - TE -2 0 0
## 4 EC - TS 3 0 0
## 5 TB - TS -2 0 1
## 6 TE - TS 0 1 1
## 7 EC - TT 4 0 0
## 8 TB - TT -2 0 1
## 9 TE - TT 1 1 1
## 10 TS - TT 0 1 1DTMIAd.Comparison <- DTMIAd$res$Comparison
DTMIAd.Comparison## [1] EC - TB EC - TE TB - TE EC - TS TB - TS TE - TS EC - TT TB - TT
## [9] TE - TT TS - TT
## 10 Levels: EC - TB EC - TE EC - TS EC - TT TB - TE TB - TS ... TS - TTDTMIAd.Padj <- round(DTMIAd$res$P.adj, digits=5)
DTMIAd.Padj## [1] 0 0 0 0 1 1 0 1 1 1KrusData = data.frame(DTMIAd.Comparison, DTMIAd.Padj)
KrusData## DTMIAd.Comparison DTMIAd.Padj
## 1 EC - TB 0
## 2 EC - TE 0
## 3 TB - TE 0
## 4 EC - TS 0
## 5 TB - TS 1
## 6 TE - TS 1
## 7 EC - TT 0
## 8 TB - TT 1
## 9 TE - TT 1
## 10 TS - TT 1RND = nrow(DTMIAd$res)
RND ## [1] 10krustable=kable(KrusData[1:RND,])
krustable| DTMIAd.Comparison | DTMIAd.Padj |
|---|---|
| EC - TB | 0 |
| EC - TE | 0 |
| TB - TE | 0 |
| EC - TS | 0 |
| TB - TS | 1 |
| TE - TS | 1 |
| EC - TT | 0 |
| TB - TT | 1 |
| TE - TT | 1 |
| TS - TT | 1 |
DTMIAd$dtres## [1] " Kruskal-Wallis rank sum test"
## [2] ""
## [3] "data: x and g"
## [4] "Kruskal-Wallis chi-squared = 29.3877, df = 4, p-value = 0"
## [5] ""
## [6] ""
## [7] " Comparison of x by g "
## [8] " (Bonferroni) "
## [9] "Col Mean-|"
## [10] "Row Mean | EC TB TE TS"
## [11] "---------+--------------------------------------------"
## [12] " TB | 5.264338"
## [13] " | 0.0000*"
## [14] " |"
## [15] " TE | 3.020269 -2.244068"
## [16] " | 0.0253* 0.2483"
## [17] " |"
## [18] " TS | 3.496750 -1.767587 0.476480"
## [19] " | 0.0047* 0.7713 1.0000"
## [20] " |"
## [21] " TT | 3.588972 -1.675365 0.568702 0.092221"
## [22] " | 0.0033* 0.9386 1.0000 1.0000"
## [23] ""
## [24] "alpha = 0"
## [25] "Reject Ho if p <= alpha"Resultados
El siguiente codigo da una respuesta u otra dependiendo si el valor de P calculado por la prueba de Kruskal-Wallis es mayor o menor a 0.05.
if (KTMIAd$p.value>0.05) {
dif="No existe diferencia significativa entre los tratamientos (P<0.05)"
krustable="0"
} else {
dif="Existe diferencia significativa entre los tratamientos (P<0.05)."
krustable=kable(KrusData[1:RND,])
}
dif## [1] "Existe diferencia significativa entre los tratamientos (P<0.05)."krustable| DTMIAd.Comparison | DTMIAd.Padj |
|---|---|
| EC - TB | 0 |
| EC - TE | 0 |
| TB - TE | 0 |
| EC - TS | 0 |
| TB - TS | 1 |
| TE - TS | 1 |
| EC - TT | 0 |
| TB - TT | 1 |
| TE - TT | 1 |
| TS - TT | 1 |
Existe diferencia significativa entre los tratamientos (P<0.05).
Reporte de resultados
Con los datos almacenados en variables podemos hacer una tabla como la siguiente.
| Germ | Promedio ± Error | Dif |
|---|---|---|
| EC: | 31517 ± 7491 | a |
| TB: | 65 ± 25 | b |
| TE: | 360 ± 106 | b |
| TS: | 1328 ± 538 | b |
| TT: | 220 ± 45 | b |
Graficos generados por R
También se pueden incluir graficos. Por ejemplo, la función boxplot.
boxplot(Sub.Eggs.MIAd$EggsP ~ Sub.Eggs.MIAd$Nger, main = "MIAd", xlab = "Germplasm", ylab = "Eggs per plant")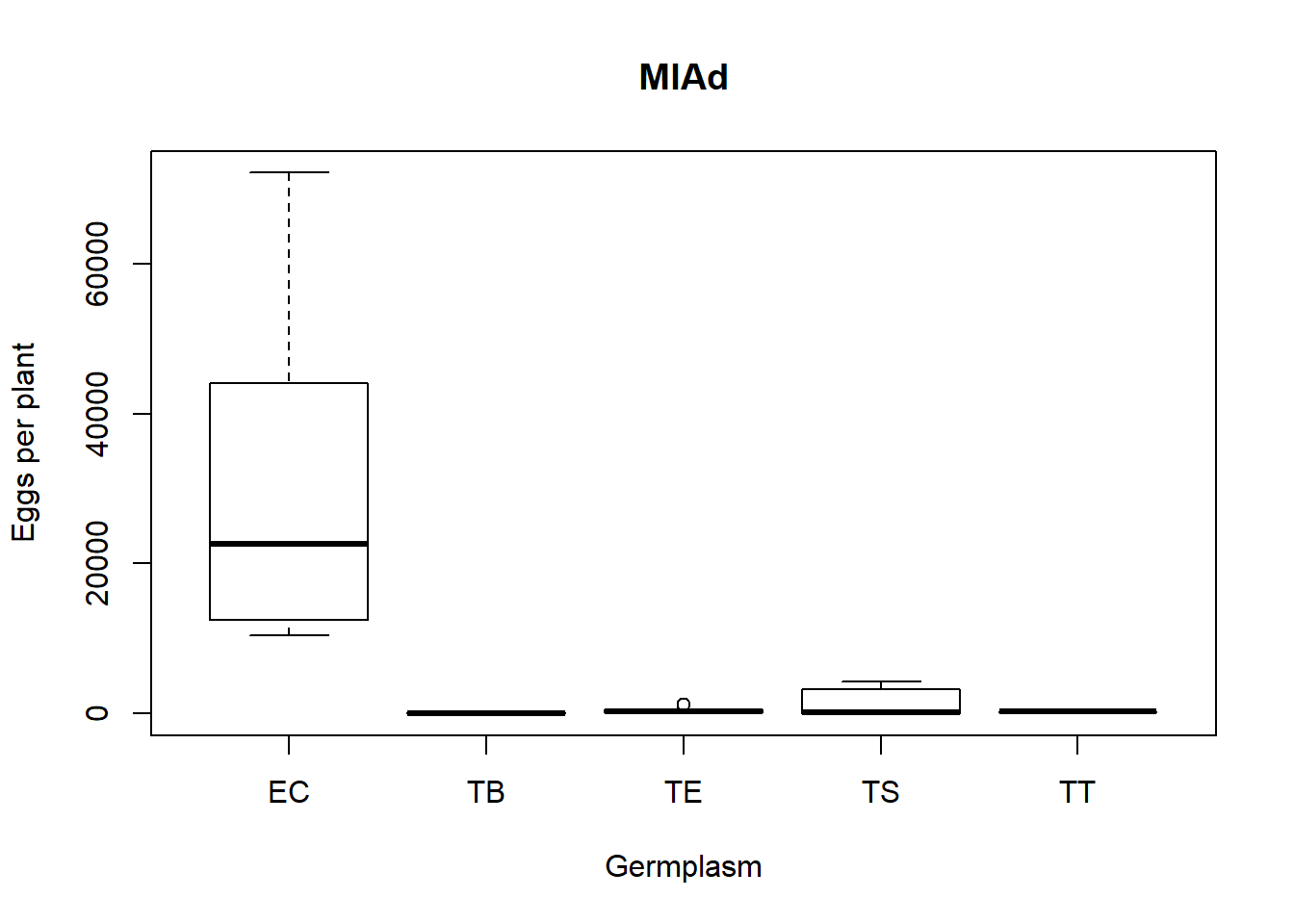
Graficos no generados por R
Si se desea incluir graficos que no son generados por codigo R, puedes usar la función knitr::include_graphics(), la cual te da mas control sobre los atributos de la imagen que la sintaxis… (por ejemplo, puedes especificar el ancho de la figura mediante out.width). La figura 1 provee un ejemplo de ello.
knitr::include_graphics(rep('/images/nuevas/20201205_Prueba_RMD01.png', 1)) ## codigo para Hugo
Figure 1: Figure caption
El codigo anterior permite generar el siguiente documento en formato PDF.
knitr::include_url(rep("https://drive.google.com/file/d/1WVU3wqwyk9sTKjaFwtysoLS3uFDiyrae/preview", 1))Figure 2: Figure caption